Kurz und Knapp
- Unsere Zellen bestehen unter anderem aus Proteinen. Die Proteinzusammensetzung hängt dabei von der Funktion und dem Zustand der jeweiligen Zelle ab.
- Mit der Massenspektrometrie (MS) lassen sich Identität und Menge der in einer Probe gefundenen Proteine ermitteln – die Analyse der Daten ist jedoch zeitintensiv.
- Das Max-Planck-Institut für Biochemie hat es geschafft, die Analyse dank KI zu beschleunigen. Dabei sind sie auch auf neue chemische Muster in Proteinen gestoßen.
KI-basiertes Programm zieht Rückschlüsse und erzeugt neues Wissen
Die letzte Station für alte Autos ist oft der Schrottplatz. Dort werden die Fahrzeuge in ihre noch verwertbaren Einzelteile zerlegt. Eine erfahrene Fachkraft des Schrottplatzes kann anschließend durch einen Blick auf die zerlegten Teile erkennen, um welchen Fahrzeugtyp es sich handelt. Vergleichbar läuft es bei Massenspektrometer (MS) ab: Um die in einer Probe enthaltenen Proteine identifizieren und quantifizieren zu können, werden zunächst die Proteine in kleinere Fragmente, die Peptide, zerlegt. Die Informationen über Identität und Menge der Peptide lassen sich als Spektren erfassen. Um die Informationen über die Proteine aus der analysierten Probe zu rekonstruieren, werden die Merkmale dieser Spektren dann mit früher erfassten Proteinbibliotheken verglichen.
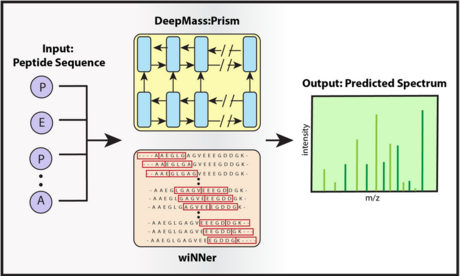
Die Forschungsgruppe des MPIB entwickelte gemeinsam mit Verily das Modell DeepMass:Prism, um die Interpretation von MS- Daten zu vereinfachen. Mittels maschinellen Lernens werden Deep Learning Algorithmen trainiert, Proteine in MS-Spektren zu übersetzen. Vergleichbare Ansätze werden auch bei der automatischen Übersetzung von Sprachen angewandt. Allerdings ist DeepMass:Prism darin geschult, Übersetzungen zwischen Proteinen und Spektren zu erstellen.
DeepMass:Prism kann vor allem dabei helfen Krankheiten zu diagnostizieren: Blutproben von Patienten sind grundsätzlich durch eine ähnliche Proteinzusammensetzung charakterisiert. Für gezielte Krankheitsdiagnosen ist es jedoch wichtig, veränderte Proteingehalte zu erkennen. Dabei unterscheiden sich die Zelltypen des menschlichen Körpers nicht nur durch das Vorkommen unterschiedlicher Proteine, sondern auch durch den unterschiedlichen Gehalt der identifizierten Proteine. Ähnlich wie beim Schrottplatz: Werden Fahrzeuge in Einzelteile zerlegt, gleichen sich die Haufen mit den Komponenten ziemlich stark. Erst die Anzahl bestimmter Teile gibt weitere Informationen. Gibt es zum Beispiel nur drei Reifen, ist von einem möglichen Defekt auszugehen. Das gleiche Prinzip lässt sich auf die Analyse von Zellen anwenden. Krankheiten können bewirken, dass bestimmte Proteine in größeren oder geringeren Mengen vorhanden sind. Das KI-basierte Programm ist in der Lage, diese individuellen Unterschiede in der Proteinmenge zu erkennen - und das zeit- und ressourcensparend. Denn anstatt die Referenzbibliotheken, mit denen die Proben verglichen werden, experimentell zu bestimmen, kann das Programm diese nun vorhersagen.
Dabei entdeckte DeepMass:Prism neue chemische Regeln, die bestimmen, wie die Peptide in kleinere Fragmente zerlegt werden. Die Vorhersagen durch DeepMass:Prism haben die Identifizierung einer neuen Art der Interaktion innerhalb von Proteinen ermöglicht. In Zukunft soll das Modell dabei helfen, neue Diagnoseinstrumente zu entwickeln.
31.05.2019
Weitere Informationen

in Kooperation mit dem